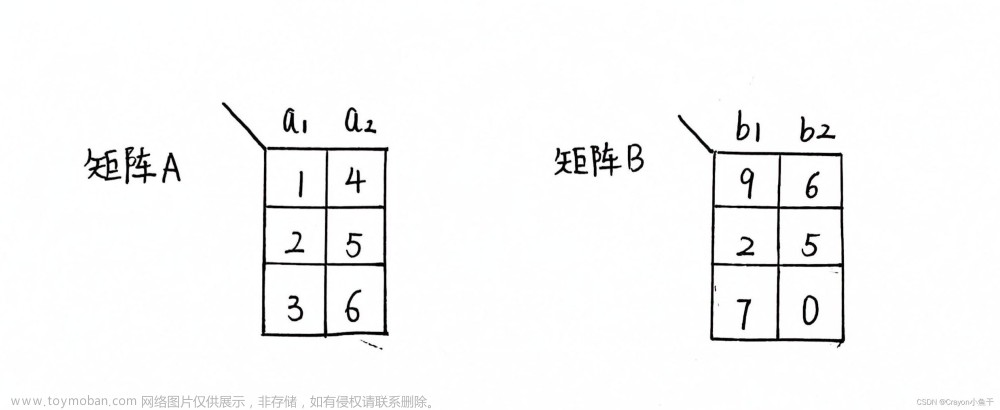
使用场景
-
1.已经确定研究的基因,但是想探索他潜在的功能,可以通过跟这个基因表达最相关的基因来反推他的功能,这种方法在英语中称为guilt of association,协同犯罪。
-
2.我们的注释方法依赖于TCGA大样本,既然他可以注释基因,那么任何跟肿瘤相关的基因都可以被注释,包括长链非编码RNA
下面操作开始:
1.加载已经整理好的癌症数据
-
load(file = "exprSet_arrange.Rdata") -
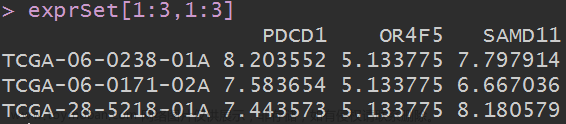
exprSet[1:3,1:3]
这个数据依然是行是样本,列是基因。
2.批量相关性分析
将第一行目的基因跟其他行的编码基因批量做相关性分析,得到相关性系数以及p值 需要大概30s左右的时间。
-
y <- as.numeric(exprSet[,"PDCD1"]) colnames <- colnames(exprSet) cor_data_df <- data.frame(colnames) for (i in 1:length(colnames)){ test <- cor.test(as.numeric(exprSet[,i]),y,type="spearman") cor_data_df[i,2] <- test$estimate cor_data_df[i,3] <- test$p.value } names(cor_data_df) <- c("symbol","correlation","pvalue")
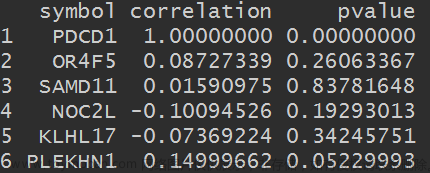
查看这个数据结构
-
head(cor_data_df)
3.筛选最相关的基因
筛选p值小于0.05,按照相关性系数绝对值选前500个的基因, 数量可以自己定
-
library(dplyr) -
library(tidyr) -
cor_data_sig <- cor_data_df %>% -
filter(pvalue < 0.05) %>% -
arrange(desc(abs(correlation)))%>% -
dplyr::slice(1:500)
4.随机选取正的和负的分别作图验证
用到的方法在以前的图有毒系列里面 图有毒系列之二
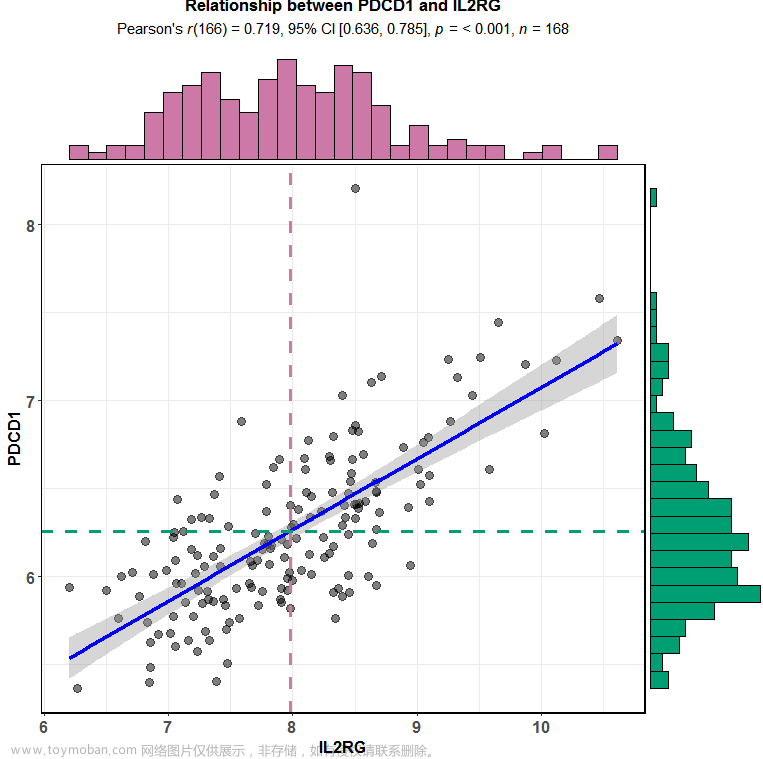
正相关的选取IL2RG
-
library(ggstatsplot) -
ggscatterstats(data = exprSet, -
y = PDCD1, -
x = IL2RG, -
centrality.para = "mean", -
margins = "both", -
xfill = "#CC79A7", -
yfill = "#009E73", -
marginal.type = "histogram", -
title = "Relationship between PDCD1 and IL2RG")
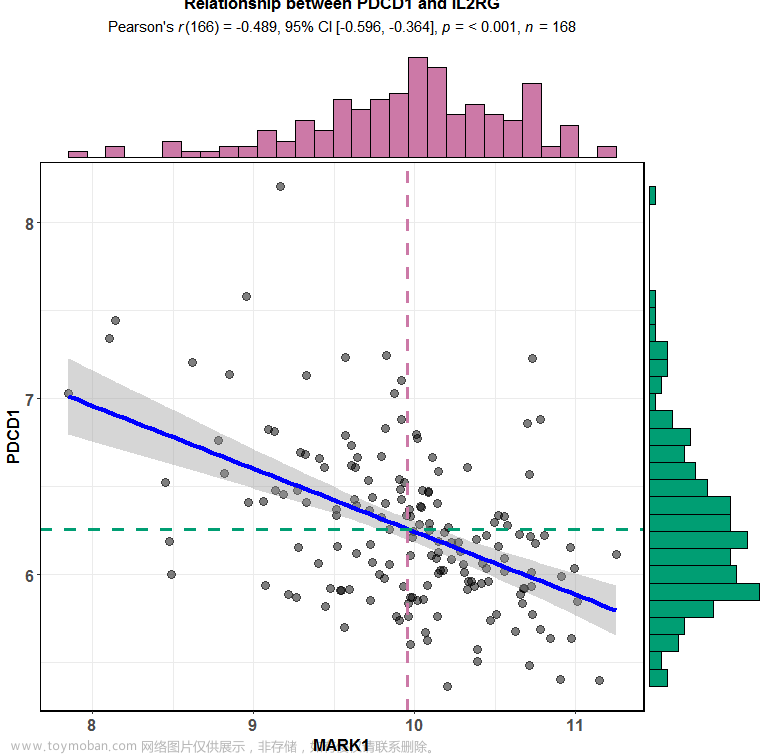
负相关的选取MARK1
-
library(ggstatsplot) -
ggscatterstats(data = exprSet, -
y = PDCD1, -
x = MARK1, -
centrality.para = "mean", -
margins = "both", -
xfill = "#CC79A7", -
yfill = "#009E73", -
marginal.type = "histogram", -
title = "Relationship between PDCD1 and IL2RG")
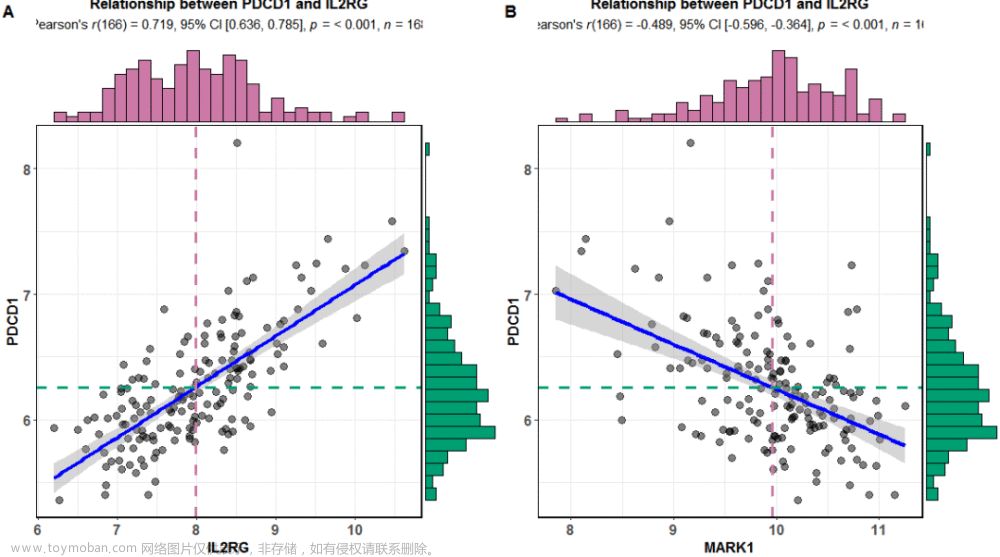
我们还可以用cowplot拼图
-
library(cowplot) p1 <- ggscatterstats(data = exprSet, y = PDCD1, x = IL2RG, centrality.para = "mean", margins = "both", xfill = "#CC79A7", yfill = "#009E73", marginal.type = "histogram", title = "Relationship between PDCD1 and IL2RG") p2 <- ggscatterstats(data = exprSet, y = PDCD1, x = MARK1, centrality.para = "mean", margins = "both", xfill = "#CC79A7", yfill = "#009E73", marginal.type = "histogram", title = "Relationship between PDCD1 and IL2RG") plot_grid(p1,p2,nrow = 1,labels = LETTERS[1:2])
setwd("/home/data/t040413/ipf/gse135893_20_PF_10_control_scRNAseq")
getwd()
#install.packages("ggside") #.libPaths(c("/home/data/t040413/R/yll/usr/local/lib/R/site-library", "/home/data/t040413/R/x86_64-pc-linux-gnu-library/4.2", "/usr/local/lib/R/library"))
.libPaths(c("/home/data/t040413/R/yll/usr/local/lib/R/site-library", "/home/data/t040413/R/x86_64-pc-linux-gnu-library/4.2", "/usr/local/lib/R/library"))
library(ggstatsplot)
load("/home/data/t040413/ipf/gse135893_20_PF_10_control_scRNAseq/mydata_for_gpx3_ecm_association.rds")
head(mydata)
ggscatterstats(data =mydata,
y = ECM_Score,
x = GPX3,
centrality.para = "mean",
margins = "both",
xfill = "#CC79A7",
yfill = "#009E73",
marginal.type = "histogram",
title = "Relationship between GPX3 and ECM_Score from fibroblasts in GSE135895")
.libPaths(c("/home/data/refdir/Rlib",
"/home/data/t040413/R/x86_64-pc-linux-gnu-library/4.2",
"/usr/local/lib/R/library"))5.下面进行聚类分析
既然确定了相关性是正确的,那么我们用我们筛选的基因进行富集分析就可以反推这个基因的功能
-
library(clusterProfiler) -
#获得基因列表 -
library(stringr) -
gene <- str_trim(cor_data_sig$symbol,'both') -
#基因名称转换,返回的是数据框 -
gene = bitr(gene, fromType="SYMBOL", toType="ENTREZID", OrgDb="org.Hs.eg.db") -
go <- enrichGO(gene = gene$ENTREZID, OrgDb = "org.Hs.eg.db", ont="all")
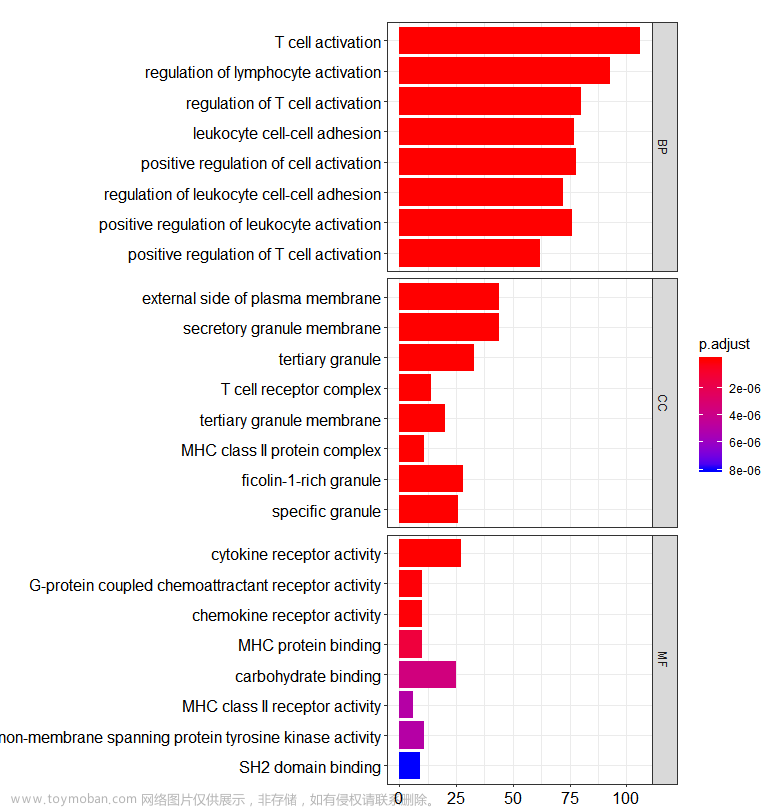
这里因为是计算的所有GO分析的三个分类,所以可以合并作图
这是条形图
-
barplot(go, split="ONTOLOGY")+ facet_grid(ONTOLOGY~., scale="free")
这是气泡图
-
dotplot(go, split="ONTOLOGY")+ facet_grid(ONTOLOGY~., scale="free")
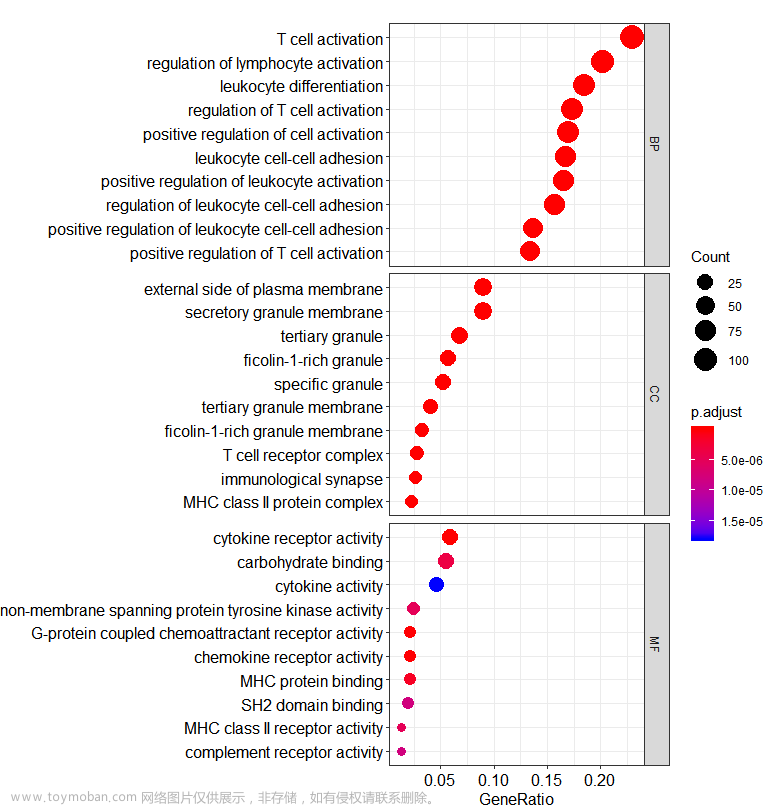
这时候,我们能推断PDCD1这个基因主要参与T细胞激活,细胞因子受体活性调剂等功能,大致跟她本身的功能是一致的。
这种方法,即使是非编码基因也可以注释出来,想到长链非编码基因的数量,真是钱途无量。
 文章来源:https://www.toymoban.com/news/detail-427928.html
文章来源:https://www.toymoban.com/news/detail-427928.html
欢迎关注微信:生信小博士 文章来源地址https://www.toymoban.com/news/detail-427928.html
到了这里,关于表达矩阵任意两个基因相关性分析 批量相关性分析 tcga geo 矩阵中相关性强的基因对 基因相关性 ecm matrisome与gpx3的文章就介绍完了。如果您还想了解更多内容,请在右上角搜索TOY模板网以前的文章或继续浏览下面的相关文章,希望大家以后多多支持TOY模板网!