目录
一、准备数据
1.查看数据
二、数据探索性分析
1.数据描述型分析
2.各特征值与结果的关系
a)研究各个特征值本身类别
b)研究怀孕次数特征值与结果的关系
c)其他特征值
3.研究各特征互相的关系
三、数据预处理
1.去掉唯一属性
2.处理缺失值
a)标记缺失值
b)删除缺失值行数
c)用合理值代替缺失值
3.异常值处理
四、机器学习(分类模型)
1.决策树
a)建立决策树模型
b)模型评估
c)参数优化
d)重新建立模型
e)决策树可视化
2.贝叶斯
3.神经网络
a)sklearn神经网络
b)Tensorflow学习神经网络
c)准确率与损失值可视化
4.三个模型总体一览
五、错误与总结
1.相关性分析
2.异常值处理问题
3.贝叶斯与神经网络模型的优化问题
六、参考资料
七、源码
一、准备数据
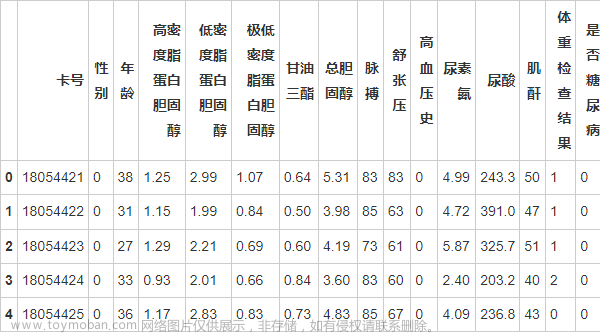
1.查看数据
皮马印第安人糖尿病数据集(Pima Indians Diabetes Dataset)
https://blog.csdn.net/weixin_39877898/article/details/110690932
皮马印第安人糖尿病数据集涉及基于给定的医疗措施预测皮马印第安人5年内糖尿病的发病情况。这是一个二分类问题,每一类对应的观测值数量并不均衡。该数据集共有768组观测值,每组观测值有8个输入变量和1个输出变量。变量名称及含义如下所示:
| 0列为怀孕次数; |
| 1列为口服葡萄糖耐量试验中2小时后的血浆葡萄糖浓度; |
| 2列为舒张压(单位:mm Hg) |
| 3列为三头肌皮褶厚度(单位:mm) |
| 4列为餐后血清胰岛素(单位:mm) |
| 5列为体重指数(体重(公斤)/ 身高(米)^2) |
| 6列为糖尿病家系作用 |
| 7列为年龄 |
| 8列为分类变量(0或1) |

二、数据探索性分析
1.数据描述型分析

从最小值可以判断出肯定存在无意义的零值作为缺失值处理

2.各特征值与结果的关系
a)研究各个特征值本身类别
研究各个特征值类别,遍历数据集每个特征值







b)研究怀孕次数特征值与结果的关系


本来应该生成17个图,但由于缺失值未处理,以及怀孕次数特征值中有部分类别完全为1或者0,因此有部分饼图绘制不出。因此会有报错,但是没有问题的特征值图照常显示。
c)其他特征值
由于该数据类型大部分为数值型数据,因此其他特征值的饼图不具备参考意义所以没有进行绘制饼图实验。
3.研究各特征互相的关系
a)相关性研究
根据相关性图的研究,不难看出各个指标相关性并未重复相关,因此在这里不选择去掉某一属性


b)关系矩阵图

三、数据预处理
1.去掉唯一属性
无唯一属性值
2.处理缺失值
a)标记缺失值
查看是否存在缺失值


具体来说,以下列具有无效的零最小值,输出无效零值


标记缺失值

查看是否已标记缺失值

标记成功
b)删除缺失值行数
对于缺失值少的删除其所在行(Glucose)(BMI)

得到如下结果:

c)用合理值代替缺失值
对于缺失值较多的用合理值代替缺失值(Pregnancies)(BloodPressure(SkinThickness)(Insulin)



最终处理结果(data1)

3.异常值处理
查看是否存在异常值

判断应该不存在异常值,原因是该数据集中异常值判断需要专业的医学知识,因此判断不出异常值

四、机器学习(分类模型)
1.决策树
a)建立决策树模型
首先先将数据集分为训练集与测试集,按照七比三分

接着开始建立决策树模型

b)模型评估
参数评估得到准确率

c)参数优化
通过网格搜索函数
可以看见最优准确率为:0.8256813602634037
最优参数为:
{'criterion': 'gini',
'max_depth': 6,
'min_impurity_decrease': 0.0,
'min_samples_leaf': 26,
'splitter': 'best'}

d)重新建立模型
参数优化了0.03

e)决策树可视化
导入相关函数,可视化决策树

在小黑框中将dot文件解码成png文件

结果如下

2.贝叶斯

3.神经网络
a)sklearn神经网络

b)Tensorflow学习神经网络
(此处属于进一步学习,参照了B站基于神经网络的垃圾分类 v.2021 - 人工智能垂直领域工程项目案例库)
训练过程
第一步开始进行100次训练

结果展示

第100次训练的时候准确率为0.7243,测试集的准确率为0.7345

c)准确率与损失值可视化
测试集准确率与训练集准确率相差不大,因此模型训练没有出现问题
先绘制可视化训练集和测试集准确率变化情况图

结果如下图所示:

再绘制可视化训练集和测试集损失值变化情况图

结果如下图所示:

4.三个模型总体一览

五、错误与总结
1.相关性分析
相关性这里其实是有少数几个属性相关性达到了0.60以上,严谨做法应该删掉其中一个属性,但由于时间关系并未进行深入研究,因此并未对这几个属性进行深入研究
2.异常值处理问题
从学术意义上来讲,异常值的判定要与数据基本属性匹配,在该糖尿病数据集中作为非医学类学生不具备相应的知识储备,因此异常值仅仅只能表示出来,无法判断数据中异常值的存在。所以下一个实验也会有倾向性的选择数据集
3.贝叶斯与神经网络模型的优化问题
没有找到相关资料去优化贝叶斯与神经网络模型,因此无法将该两个模型做进一步准确率优化
六、参考资料
决策树参考:决策树系列(四)——基于决策树算法实现员工离职率预测_初一的博客-CSDN博客
鸢尾花示例:鸢尾花(iris)数据集分析 - 简书
数据预处理:How to Handle Missing Data with Python
神经网络深度学习:
【开源】基于神经网络的垃圾分类 v.2021 - 人工智能垂直领域工程项目案例库_哔哩哔哩_bilibili
七、源码
# 导入相关包
import numpy as np
import pandas as pd
from numpy import nan
#%matplotlib inline
import matplotlib.pyplot as plt
plt.style.use('seaborn')
import seaborn as sns
sns.set_style("whitegrid")
#决策树
from sklearn.tree import DecisionTreeClassifier as DTC
from sklearn.tree import export_graphviz
#贝叶斯
from sklearn.naive_bayes import GaussianNB
#神经网络
from sklearn.neural_network import MLPClassifier
#统计
from sklearn.impute import SimpleImputer
from sklearn.model_selection import GridSearchCV
from sklearn import metrics
from sklearn.model_selection import train_test_split
#Tensorflow神经网络
from keras.models import Sequential
from keras.layers.core import Dense, Activation
# 导入数据集
#查看数据
diabetes = pd.read_csv('D:\数据挖掘\实验一\diabets.csv')
diabetes.info()
#print(diabetes.describe())
#数据探索
#直方图
for k in ['Pregnancies','Glucose','BloodPressure','SkinThickness','Insulin','BMI']:
plt.figure(figsize=(15,5))
sns.barplot(diabetes[k]. value_counts().index,diabetes[k].value_counts().values)
plt.title(k)
#饼图
#绘制饼图
#此处由于数据特性需要手动设置断点
#怀孕次数与是否患糖尿病的关系
fig = plt.figure(figsize=(25,15))#建立图像
l1 = list(diabetes['Pregnancies'].unique())
for i,j in enumerate(l1):
ax = fig.add_subplot(5,4,i+1)
p = diabetes[diabetes['Pregnancies'] == j]['Outcome'] .value_counts() #计算怀孕次数J特征值所对应的目标特征outcome的特征值数量
ax.pie(p,labels=['no','yes'],autopct='%0.2f%%',explode=(0,0.2)) #绘画怀孕次数各个特征值
ax.set_title(j)
plt.show()文章来源地址https://www.toymoban.com/news/detail-503211.html
#各特征值与结果的关系
# 设置颜色主题
antV = ['#1890FF', '#2FC25B', '#FACC14', '#223273', '#8543E0', '#13C2C2', '#3436c7', '#F04864']
#生成各特征之间的关系矩阵图
g = sns.pairplot(data=diabetes, palette=antV)
# 查看特征间的相关性
pearson_mat = diabetes.corr(method='spearman')
plt.figure(figsize=(15,15))
ax = sns.heatmap(pearson_mat,square=True,annot=True,cmap='YlGnBu')
bottom, top = ax.get_ylim()
ax.set_ylim(bottom + 0.5, top - 0.5)
plt.show()
#数据预处理
#缺失值处理
# 按列统计零的值
num_missing = (diabetes == 0).astype(int).sum(axis=0)
# 输出结果
print(num_missing)
#用NAN标记0值
diabetes[['Pregnancies','Glucose','BloodPressure','SkinThickness','Insulin','BMI']] = diabetes[['Pregnancies','Glucose','BloodPressure','SkinThickness','Insulin','BMI']].replace(0, nan)
print(diabetes.head(20))
#处理缺失值少的特征值删除这两列中缺失值对应的行数
data = diabetes.dropna(axis=0,subset = ["Glucose", "BMI"])
print(data)
#查看删除缺失值后的缺失值统计
print(data.isnull().sum())
# 用平均列值填充缺少的值
values = data.values
#定义插补器
imputer = SimpleImputer(missing_values=nan, strategy='mean')
#转换数据集
datas = imputer.fit_transform(values)
data1 = pd.DataFrame(datas)
#查看补足缺失值后的缺失值统计
data1_num_missing = (data1 == 0).astype(int).sum(axis=0)
print(data1_num_missing)
data1.info()
#异常值处理
# 异常值处理
numeric_columns = []
object_columns = []
for c in data1.columns[0:-1]:
if data1[c].dtype == 'object':
object_columns.append(c)
else:
numeric_columns.append(c)
fig = plt.figure(figsize=(20,20))
for i,col in enumerate(numeric_columns):
ax = fig.add_subplot(5,3,i+1)
sns.boxplot(data1[col],orient='v',ax=ax)
plt.xlabel(col)
plt.show()
#数据挖掘
#数据拆分为测试集与训练集
# 载入特征和标签集
X = data1[[0,1,2,3,4,5,6,7]]
y = data1[8]
#将数据集按照7:3的比例进行拆分
train_X, test_X, train_y, test_y = train_test_split(X, y, test_size = 0.3, random_state = 101)
print(train_X.shape, train_y.shape, test_X.shape, test_y.shape)
#检测不同模型的准确性
#决策树
#建立决策树模型,基于信息熵
dtc = DTC(criterion='entropy')
dtc.fit(train_X, train_y) #训练模型
prediction = dtc.predict(test_X)
#此模型计算的准确率
print('The accuracy of the Decision Tree is: {0}'.format(metrics.accuracy_score(prediction,test_y)))
l2 = ['Pregnancies','Glucose','BloodPressure','SkinThickness','Insulin','BMI','DiabetesPedigreeFunction','Age']
#[*zip(l2,dtc.feature_importances_)]#显示每个特征值的重要性
# 使用网格搜索寻找最优参数对模型进行优化
gini_thresholds = np.linspace(0,0.5,20)
parameters = {
'splitter':('best','random')
,'criterion':("gini","entropy")
,"max_depth":[*range(1,10)]
,'min_samples_leaf':[*range(1,50,5)]
,'min_impurity_decrease':[*np.linspace(0,0.5,20)]
}
clf = DTC(random_state=25)
GS = GridSearchCV(clf, parameters, cv=10,scoring='roc_auc')
GS.fit(train_X, train_y)
print(GS.best_score_)
print(GS.best_params_)
#重新建立模型
dtc_1 = DTC(
criterion ='gini',
max_depth = 6,
min_impurity_decrease=0.0,
min_samples_leaf = 26,
splitter = 'best'
)
dtc_1.fit(train_X, train_y) #训练模型
prediction = dtc_1.predict(test_X)
#此模型计算的准确率
print('The accuracy of the Decision Tree is: {0}'.format(metrics.accuracy_score(prediction,test_y)))
#导入相关函数,可视化决策树
x = pd.DataFrame(data1)
with open('D:/数据挖掘/实验一/diabetes.dot','w',encoding='utf-8')as f:
f = export_graphviz(dtc,feature_names=l2,class_names=['0', '1'],out_file=f,rounded=True)
#贝叶斯
clf = GaussianNB()
clf.fit(train_X, train_y)
prediction = clf.predict(test_X)
#获取两个类别的先验概率
print('两个类别的先验概率:',clf.class_prior_ )
#此模型计算的准确率
print('The accuracy of the NaiveBayes is{0}'.format(metrics.accuracy_score(prediction,test_y)))
#神经网络
#sklearn学习神经网络
mlp=MLPClassifier(solver='lbfgs',alpha=1e-5,hidden_layer_sizes=(10,5))
mlp.fit(train_X, train_y)
prediction = mlp.predict(test_X)
print('The accuracy of the neural network is{0}'.format(metrics.accuracy_score(prediction,test_y)))
#Tensorflow学习神经网络
#建立模型
mlp1 = Sequential()
# 通过.add()方法一个个的将layer(层)加入模型中
mlp1.add(Dense(input_dim = 8,units = 7)) # Dense表示全连接层 units为隐藏层的单元数
mlp1.add(Activation('relu')) # 用relu函数作为激活函数,能够大幅度提高准确度
mlp1.add(Dense(input_dim = 7,units = 1))
mlp1.add(Activation('sigmoid')) # 由于所示0-1输出,用sigmoid函数作为激活函数
# 编译 优化器optimizer,损失函数loss,指标列表metrics(性能评估)
mlp1.compile(loss = 'binary_crossentropy',optimizer = 'adam',metrics = ['accuracy'])
#准确率与损失值可视化
# 训练模型,学习100次 batch_size就是每批处理的样本的个数
history_fit = mlp1.fit(train_X, train_y,epochs = 100,batch_size = 10,validation_data = (test_X,test_y))
# 将训练过程的中间信息保留为json文件
with open(r'D:/数据挖掘/实验一/history_fit.json',"w") as json_file:
json_file.write(str(history_fit))
# 查看训练集准确率变化
acc = history_fit.history['accuracy']
# 查看测试集准确率变化
val_acc = history_fit.history['val_accuracy']
# 查看训练集损失值变化
loss = history_fit.history['loss']
# 查看测试集损失值变化
val_loss = history_fit.history['val_loss']
# 可视化训练集和测试集准确率变化情况
epochs = range(1,len(acc) + 1)
plt.figure("acc")
plt.plot(epochs,acc,'r-',label = 'Training acc')
plt.plot(epochs,val_acc,'b',label = 'validation acc')
plt.title("The comparision of train_acc and val_acc")
plt.legend()文章来源:https://www.toymoban.com/news/detail-503211.html
plt.show()
# 可视化训练集和测试集损失值变化情况
plt.figure("loss")
plt.plot(epochs,loss,'r-',label = 'Training loss')
plt.plot(epochs,val_loss,'b',label = 'validation loss')
plt.title("The comparision of train_loss and val_loss")
plt.legend()
plt.show()
到了这里,关于数据挖掘-实战记录(一)糖尿病python数据挖掘及其分析的文章就介绍完了。如果您还想了解更多内容,请在右上角搜索TOY模板网以前的文章或继续浏览下面的相关文章,希望大家以后多多支持TOY模板网!