生成式预训练模型在自然语言处理和计算机视觉等各个领域取得了显著的成功。特别是将大规模多样化的数据集与预训练的Transformer相结合,已经成为开发基础模型的一种有前途的方法。文本由单词组成,细胞可以通过基因进行表征。这种类比启发作者探索细胞和基因生物学基础模型的潜力。通过利用呈指数增长的单细胞测序数据,作者首次尝试通过对超过1000万个细胞进行生成式预训练来构建单细胞基础模型。scGPT有效地捕捉了有关基因和细胞的生物学见解。此外,该模型可以轻松进行微调,以在各种下游任务中实现最先进的性能,包括批次整合、多组学整合、细胞类型注释、基因网络推断。
来自:scGPT: Towards Building a Foundation Model for Single-Cell Multi-omics Using Generative AI
背景
生成式预训练模型最近在许多领域取得了前所未有的成功。这些基础模型可以轻松地适应各种下游任务和场景。与从头开始训练的特定任务模型相比,预训练模型在多个任务上展示出了更好的性能。这显示了它们在这些领域具有任务无关和深入理解知识的证据。尽管基础模型在其他领域取得了显著成功,但目前基于机器学习的单细胞研究相对分散。由于测序能力和研究问题的范围限制,每项研究中使用的数据集的广度和规模往往有限。这就需要一个在大规模数据上进行预训练的基础模型,以实现对单细胞生物学的全面理解。在此项研究中,作者通过对超过1000万个细胞进行生成式预训练,首次尝试构建了一个单细胞基础模型,即scGPT。作者证明了预训练模型在基因和细胞层面捕捉到了有意义的生物学见解。在零样本学习中,预训练模型能够在未见过的数据集上揭示有意义的细胞簇。通过在少样本学习设置中进行微调,模型在各种下游任务上取得了最先进的性能,包括批次校正、多组学整合、细胞类型注释、伪细胞生成和基因网络推断。预训练基础模型将进一步增进我们对细胞生物学的理解,并为未来的新发现奠定基础。
方法
单细胞测序可以捕获细胞水平的遗传特征。例如,单细胞RNA测序(scRNA-seq)可以测量基因表达水平,从而揭示细胞的身份、状态和功能。最近总结的细胞参考图谱,如人类细胞图谱,包含来自不同器官和组织的数百万个单细胞,提供了对细胞异质性的表征。作者引入了scGPT作为单细胞领域的生成预训练基础模型。核心模型采用了堆叠的Transformer层和多头注意力机制,可以同时学习细胞和基因的特征表示。
图1A展示了一个包括预训练和微调两阶段的工作流程。在预训练阶段,作者从CellXGene门户网站收集了超过1030万个来自血液和骨髓细胞的scRNA-seq数据用于训练。作者引入了特别设计的注意力掩码和生成式训练流程,以自监督的方式训练scGPT模型,共同优化细胞和基因的表示。在训练过程中,模型逐渐学会根据简单的细胞或基因表达提示生成细胞的基因表达水平。在微调阶段,研究人员可以将预训练的模型应用于新的数据集和特定的任务。
scGPT通过基因表达建模从多样的单细胞数据中学习细胞和基因的表示。为了促进基因表示的学习,作者采用基因表达预测(GEP)作为生成自监督目标,以自回归的方式从已知的标记中迭代预测未知标记的基因表达值。为了增强细胞表示的学习,作者设计了基于细胞建模的基因表达预测(GEPC)目标,模型从细胞表示中预测基因表达值。这在基因表达谱和细胞异质性之间建立了直接的联系,使得在scGPT框架内可以进行联合优化。此外,scGPT的特征式架构可以轻松扩展到多种测序模态、批次。scGPT作为一个强大的单细胞特征提取器,可以在之前未见过的数据集上发挥作用。在基准实验中,scGPT优于最近的方法,并在所有下游任务中取得了最先进的结果。这证明了预训练的好处以及所学知识在不同应用场景中的可转移性。通过提供一个稳健且统一的框架,scGPT使得单细胞研究人员能够轻松地在相关研究中利用预训练的基础模型。
- 图1:模型框架。
- a:模型首先根据来自细胞图谱的大规模scRNA-seq数据进行生成训练。对于下游应用,可以在新数据上微调预训练的模型权重。scGPT的核心组件包含堆叠的Transformer块,具有专门用于生成训练的注意力掩模。可以将scGPT应用于各种任务,包括聚类、批校正、细胞类型注释、多组学整合、基因扰动预测(genetic perturbation prediction)和基因网络推断。
- b:Input embedding的视图。输入包含三层信息,gene token、表达值和condition token(模态、批次、扰动条件)。
- c:scGPT中Transformer层的视图。其中引入了一个特别设计的注意力掩模,用于对单细胞测序数据进行生成式预训练。
Input embedding
scRNA-seq被处理为矩阵 X ∈ R N × G X\in R^{N\times G} X∈RN×G,其每个元素 X i , j X_{i,j} Xi,j代表RNA的read count。这个矩阵被称为原始矩阵。输入scGPT包含三个部分:gene tokens(对于ATAC则是peak region tokens),expression values和condition tokens。
对于gene tokens,基因是scGPT的最小单位,相当于NLP中的单词,因此,将gene名称作为token,并在完整的token词汇表中分配给每个gene g j g_{j} gj一个独一的整数标识 i d ( g j ) id(g_{j}) id(gj)。这种方法提供了很大的灵活性来协调不同基因集相关的多个研究(比如由不同的测序技术或预处理管道生成)。特别的,通过在不同研究中取所有基因的并集可以将不同的gene token整合到一个公共词汇表。此外,还在词汇表中加入了特殊的标记,比如 < c l s > <cls> <cls>用于将所有基因聚合到一个细胞, < p a d > <pad> <pad>用于将输入扩展到固定长度。细胞 i i i的gene tokens被表示为 t g ( i ) ∈ N M t_{g}^{(i)}\in N^{M} tg(i)∈NM( N N N为自然数): t g ( i ) = [ i d ( g 1 ( i ) ) , i d ( g 2 ( i ) ) , . . . , i d ( g M ( i ) ) ] t_{g}^{(i)}=[id(g_{1}^{(i)}),id(g_{2}^{(i)}),...,id(g_{M}^{(i)})] tg(i)=[id(g1(i)),id(g2(i)),...,id(gM(i))]其中, M M M为预定义输入长度,通常为高变基因数。
对于expression values, X X X在作为输入前需要进行额外处理,对于细胞 i i i,处理后有 x j ( i ) x_{j}^{(i)} xj(i): x ( i ) = [ x 1 ( i ) , . . . , x M ( i ) ] x^{(i)}=[x_{1}^{(i)},...,x_{M}^{(i)}] x(i)=[x1(i),...,xM(i)]对于condition token,包含与个体基因相关的元信息,比如functional pathways(用pathways tokens表示),为了表示position-wise的condition tokens,作者使用与输入基因共享相同维度的输入向量: t c ( i ) = [ t c , 1 ( i ) , t c , 2 ( i ) , . . . , t c , M ( i ) ] t_{c}^{(i)}=[t_{c,1}^{(i)},t_{c,2}^{(i)},...,t_{c,M}^{(i)}] tc(i)=[tc,1(i),tc,2(i),...,tc,M(i)]其中, t c , j ( i ) t_{c,j}^{(i)} tc,j(i)表示与某个条件对应的整数索引。
作者使用Conv层 e m b g emb_{g} embg和 e m b c emb_{c} embc编码gene token和condition token。为了促进将每个token映射到固定长度 D D D的embedding向量,使用全连接层 e m b x emb_{x} embx增强expression values。最终细胞 i i i的embedding为 h ( i ) ∈ R M × D h^{(i)}\in R^{M\times D} h(i)∈RM×D: h ( i ) = e m b g ( t g ( i ) ) + e m b x ( x ( i ) ) + e m b c ( t c ( i ) ) h^{(i)}=emb_{g}(t_{g}^{(i)})+emb_{x}(x^{(i)})+emb_{c}(t_{c}^{(i)}) h(i)=embg(tg(i))+embx(x(i))+embc(tc(i))
scGPT
自注意力机制用于 M M M个token的序列,这可以捕捉基因之间的相互作用。堆叠的transformer为: h 0 ( i ) = h ( i ) h_{0}^{(i)}=h^{(i)} h0(i)=h(i) h l ( i ) = t r a n s f o r m e r ( h l − 1 ( i ) ) , ∀ l ∈ [ 1 , n ] h_{l}^{(i)}=transformer(h_{l-1}^{(i)}),\forall l\in[1,n] hl(i)=transformer(hl−1(i)),∀l∈[1,n]作者使用 h n ( i ) ∈ R M × D h_{n}^{(i)}\in R^{M\times D} hn(i)∈RM×D用于基因水平(比如基因表达预测GEP)和细胞水平的任务。对于细胞水平任务,首先整合 h n ( i ) h_{n}^{(i)} hn(i)到细胞向量,再用于细胞分类。
每个细胞都被认为是一个由基因组成的句子 h c ( i ) ∈ R D h_{c}^{(i)}\in R^{D} hc(i)∈RD(由 h n ( i ) h_{n}^{(i)} hn(i)聚合得到)。在scGPT中,作者使用特定的token < c l s > <cls> <cls>作为细胞表示,用于学习transformer中的pooling操作。 < c l s > <cls> <cls>被附加到input tokens的开头,并且以最终的embedding的这个位置(即 < c l s > <cls> <cls>)作为细胞表示。因此,细胞embedding h c ( i ) h_{c}^{(i)} hc(i)等于堆叠transformer的最后一层的第一行 h n ( i ) [ < c l s > ] h_{n}^{(i)}[<cls>] hn(i)[<cls>]。
作者使用额外的tokens集合来表示不同的批次和测序模态,这和condition tokens的介绍是一样的。模态的token t m ( i ) t_{m}^{(i)} tm(i)与个体输入 g j g_{j} gj相关(比如个体输入特征可以是gene,region,protein)。批次的token在细胞级别上,相同的批次token t b ( i ) t_{b}^{(i)} tb(i)被复制到细胞 i i i的每个最小特征上: t b ( i ) = [ t b , 1 ( i ) , . . . , t b , M ( i ) ] = [ C , . . . , C ] t_{b}^{(i)}=[t_{b,1}^{(i)},...,t_{b,M}^{(i)}]=[C,...,C] tb(i)=[tb,1(i),...,tb,M(i)]=[C,...,C]在前面描述的三种token与这里批次token和模态token的区别是,批次token和模态token不用作transformer的输入。相反,在输入特定的微调目标之前,它们在特征级别或细胞级别上与transformer输出拼接。这是为了防止Transformer放大相同模态特征内的注意力,而削弱不同模态的特征。此外,明确模态和批次的身份有助于下游任务的建模。当模型学习预测基于模态或批次身份的表达时,这些偏差被隐式地从基因和细胞表示本身中移除,这是一种便捷的批校正技术。
以多组学整合为例,拼接transformer输出和模态以及批次的embedding,作为下游微调目标的输入: h n ′ ( i ) = c o n c a t ( h n ( i ) , e m b b ( t b ( i ) ) + e m b m ( t m ( i ) ) ) h'^{(i)}_{n}=concat(h^{(i)}_{n},emb_{b}(t_{b}^{(i)})+emb_{m}(t_{m}^{(i)})) hn′(i)=concat(hn(i),embb(tb(i))+embm(tm(i)))其中, e m b b emb_{b} embb和 e m b m emb_{m} embm为批次和模态的embedding层。
对于批次整合,将批次embedding与细胞表示拼接,得到下游的输入为: h c ′ ( i ) = c o n c a t ( h c ( i ) , e m b b ( t b ( i ) ) ) h'^{(i)}_{c}=concat(h^{(i)}_{c},emb_{b}(t_{b}^{(i)})) hc′(i)=concat(hc(i),embb(tb(i)))
生成式预训练
在模型训练之前,表达值被归一化、对数变换。为了加快训练速度,将输入限制为每个输入细胞的非零表达基因(这就是gene token唯一性带来的优势)。该策略为随后的微调阶段提供了有用的预训练结果,在此阶段默认包括所有非零表达的基因。
自注意力被广泛用于捕获token之间的共现模式。在自然语言处理中,这主要通过两种方式实现:
- 在BERT中使用掩码token,在模型的输出中预测输入序列中的随机掩码token;
- 在Transformer解码器模型(如OpenAI GPT系列)中具有顺序预测的自回归模型。
OpenAI GPT3和GPT4中使用的生成式预训练采用统一的框架,其中模型从已知输入token组成的"提示符"(prompt)中预测最有可能的下一个token。该框架提供了很大的灵活性,可用于各种自然语言生成应用,并展示了诸如zero-shot和few-shot设置中的上下文感知新功能。作者认为生成式训练可以以类似的方式对单细胞模型有益。具体来说,对两个任务感兴趣:
- 基于已知基因表达生成未知基因表达值,即通过"gene prompts"生成;
- 在给定输入细胞类型条件下生成全基因组表达,即通过"cell prompts"生成。
尽管使用了类似的token和prompt,但由于数据的无序性质,对gene read count进行建模与自然语言本质上是不同的。与句子中的单词不同,细胞内基因的顺序是可以互换的,并且没有"下一个基因"这种概念可以预测。这使得直接在单细胞中应用GPT模型具有挑战性。为了应对这一挑战,作者为scGPT开发了一种专门的注意力机制,该机制根据注意力得分定义了预测的顺序。
scGPT的注意力掩码以统一的方式支持基因提示(gene prompts)和细胞提示(cell prompts)。将二元注意力掩码应用于Transformer块的自注意力图。对于输入 h l ( i ) ∈ R M × D h_{l}^{(i)}\in R^{M\times D} hl(i)∈RM×D( M M M个token),transformer block生成 M M M个query和key vectors去计算注意力图 A ∈ R M × M A\in R^{M\times M} A∈RM×M,注意力掩码也是 M × M M\times M M×M的。可视化的注意力mask见图S1-A,与掩码每一列相关联的token标识标注在图的底部,即 < c l s > < cls > <cls>、已知基因和未知基因。 h l ( i ) h_{l}^{(i)} hl(i)中的每个token都可以是这三组中的一组:
- 用于表示细胞的 < c l s > <cls> <cls>;
- 已知基因的token embedding;
- 未知基因(其表达值expression values需要预测);
scGPT的mask attention的法则是只允许在"已知基因"的embedding和查询基因本身之间进行注意计算。在每一代迭代中,scGPT预测一组新基因的基因表达值。这些基因依次成为下一次注意力计算迭代的"已知基因"。
如图S1-A所示,在训练过程中,从输入基因中随机选择一个比例作为未知基因,因此在输入中省略其表达值。对这些未知基因的查询,只允许对已知基因和查询基因本身进行注意力计算(比如图S1-A中的蓝色mask不是一个正方形减少的形式,而是逐渐压缩的矩形)。例如,在位置M预测的基因与细胞embedding的注意力得分,是用已知的基因和它自己,而不是其他未知的基因。推理步骤在图S1-B中说明。在细胞提示生成的推断过程中,scGPT生成了所有基于特定细胞类型的全基因组基因表达。

- 图S1:scGPT的注意力mask。
基本的微调目标
基因表达预测GEP
在每个细胞内,基因子集和对应的表达值被随机mask。scGPT可以准确预测mask位置的表达值。这种微调目标有利于模型有效编码数据集中基因之间的共表达。具体来说,使用一个全连接的MLP来估计基因 M M M在Transformer输出上的表达值。GEP优化的目标为: x ~ ( i ) = M L P ( h n ( i ) ) \widetilde{x}^{(i)}=MLP(h_{n}^{(i)}) x (i)=MLP(hn(i)) L G E P = 1 ∣ M m a s k ∣ ∑ j ∈ M m a s k c e ( x ~ j ( i ) , x j ( i ) ) L_{GEP}=\frac{1}{|M_{mask}|}\sum_{j\in M_{mask}}ce(\widetilde{x}_{j}^{(i)},x_{j}^{(i)}) LGEP=∣Mmask∣1j∈Mmask∑ce(x j(i),xj(i))其中, M m a s k M_{mask} Mmask表示掩码的位置, x ~ ( i ) ∈ N M \widetilde{x}^{(i)}\in N^{M} x (i)∈NM表示细胞 i i i的表达估计, c e ce ce为交叉熵,注意,在整合任务中,用 h n ′ ( i ) h_{n}'^{(i)} hn′(i)代替 h n ( i ) h_{n}^{(i)} hn(i)。
GEP提出了一个通用的自监督微调目标,旨在预测基因表达值。在某些下游任务中,例如扰动预测,需要预测被扰动的基因表达值,而不是原始值。把这种任务称为perturb-GEP。在perturb-GEP中,预测的表达式值被应用于所有有效的目标位置,而不仅仅是GEP中的屏蔽位置。
基于细胞的GEP(GEPC)
该目标与GEP类似,但是需要根据细胞表示 h c ( i ) h_{c}^{(i)} hc(i)来预测基因表达值,对于细胞 i i i的每个基因 j j j,创建一个query向量 q j q_{j} qj: q j = M L P ( e m b g ( t g ( i ) ) ) q_{j}=MLP(emb_{g}(t_{g}^{(i)})) qj=MLP(embg(tg(i))) x ~ j ( i ) = q j ⋅ W h c ( i ) \widetilde{x}_{j}^{(i)}=q_{j}\cdot Wh_{c}^{(i)} x j(i)=qj⋅Whc(i) L G E P C = 1 ∣ M m a s k ∣ ∑ j ∈ M m a s k c e ( x ~ j ( i ) , x j ( i ) ) L_{GEPC}=\frac{1}{|M_{mask}|}\sum_{j\in M_{mask}}ce(\widetilde{x}_{j}^{(i)},x_{j}^{(i)}) LGEPC=∣Mmask∣1j∈Mmask∑ce(x j(i),xj(i))同样的,对于整合任务,使用 h c ′ ( i ) h_{c}'^{(i)} hc′(i)代替 h c ( i ) h_{c}^{(i)} hc(i)。
细胞相似度学习ECS
这种学习可以增强细胞表示: L E C S = − ( s i m ( h c ( i ) , h c ( i ′ ) ) − β ) 2 L_{ECS}=-(sim(h_{c}^{(i)},h_{c}^{(i')})-\beta)^{2} LECS=−(sim(hc(i),hc(i′))−β)2其中, s i m sim sim表示余弦相似度函数, i i i和 i ′ i' i′为mini-batch中的两个细胞, β β β表示预定义的阈值。
细胞分类CLS
这个调优目标旨在利用学习到的细胞表示来注释细胞。使用一个单独的MLP分类器从细胞表示 h c ( i ) h_{c}^{(i)} hc(i)中预测细胞类型。该微调目标在预测的细胞类型概率和真值标签之间使用交叉熵损失进行优化。
scGPT在下游任务的微调
批次整合
在批次整合中,预先训练的基础模型与当前数据集之间的公共基因token集被保留,进一步从公共基因集中选择高变基因作为输入,基因表达值在模型训练前进行归一化、对数变换。使用预训练的模型权值初始化微调模型。所有gene token,不管表达值是zero还是non-zero,都被用于微调。上一节的基本的微调目标中的GEP,GEPC,ECS被同时优化。
细胞类型注释
对于细胞类型标注任务,在一个reference数据集上使用GT标签对模型进行了微调,并在一个外部query数据集上验证标注性能。保留了预训练的基础模型和reference集之间的公共基因token集。在模型训练之前对表达值进行预处理。使用预训练的模型权值初始化微调模型。所有表达值为zero和non-zero的基因token都用于训练。CLS微调目标用于最小化分类损失。
多组学整合
scMultiomic数据可能在每个批次中包含种不同的测序模态,这对集成分析提出了更具挑战性的方案。scMultiomic分为两种数据集成设置,配对(paired)和镶嵌(mosaic)。在配对设置中,所有样本(细胞)共享测序的所有数据模态。在镶嵌设置中,一些样本共享一些公共数据模态,但不是全部。
由于存在额外的ATAC或蛋白质token,需要从头开始训练额外的token嵌入和模型的其余部分。在训练中使用了所有同时具有zero和non-zero表达值的token。scGPT使用了额外的模态token来指示每个token的数据类型(即基因、区域,蛋白质),用于促进GEP和GEPC微调目标中的mask基因和表达值预测。在配对设置下,采用GEP和GEPC两种微调目标对模型进行优化。在mosaic设置中,DAR(Domain Adaptation via Reverse Back-propagation)被包括在内,以方便多模态批次校正。
基因网络推断
在zero-shot设置下,基于余弦相似度从scGPT模型的基因嵌入中提取基因相似度网络。在经过微调的设置中,作者以与免疫人类数据集上经过微调的scGPT模型类似的方式构建了基因网络。根据Ceglia等人的管道,进一步从包含5个或更多基因的基因嵌入簇中提取基因。
结果
scRNA-seq批次整合
在单细胞测序数据的聚类和可视化中,存在来自多个数据集或测序批次的批次效应。通过采用微调工作流程,scGPT框架有效地解决了这个挑战,通过引入定制的微调目标,可以成功纠正批次效应,同时保留了数据中固有的真实生物学信号。scGPT在批次校正后保持集成数据的生物差异方面取得了最先进的性能。
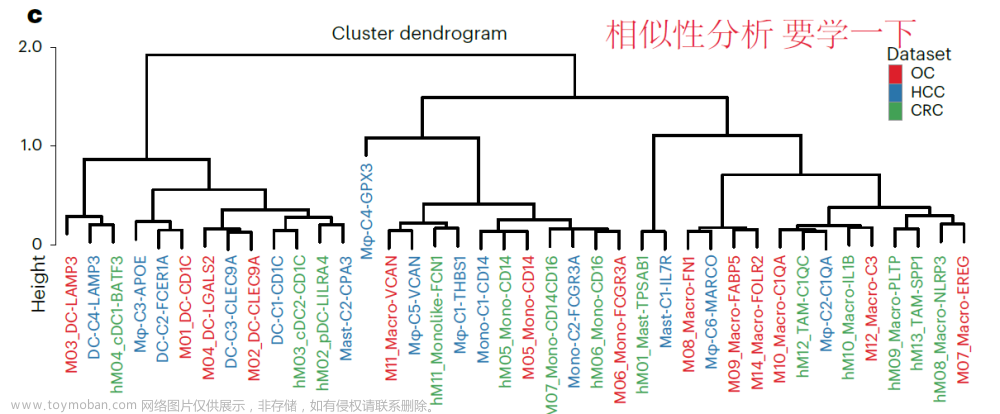
作者在两个集成数据集Immune Human(10个批次)和PBMC 10K(2个批次)上对scGPT与三种流行的集成方法scVI、Seurat和Harmony进行了基准测试。如图2A所示,在Immune Human数据集中,scGPT成功地将所有CD4+ T细胞、CD8+ T细胞和CD14+单核细胞的批次集成到各自的聚类中,而Seurat在这些细胞类型中产生了一些对应于测序批次的亚聚类。scGPT还成功将单核细胞源性树突状细胞与CD16+单核细胞分开,而scVI和Harmony两者之间这两个聚类有明显的重叠。此外,在PBMC 10K数据集中,scGPT是唯一能清楚地将细胞类型与注释的聚类分离开来的方法。相比之下,scVI、Seurat和Harmony都将此其他细胞类型与CD14+单核细胞和CD8 T细胞混淆。scGPT的优越聚类性能也体现在生物保留分数上,其中scGPT的AvgBIO分数为0.812,比Seurat和Harmony高5%,比另一种深度学习方法scVI高10%。在图2C中,scGPT在所有细胞类型聚类指标中都表现出竞争力,这归功于其生物保留性。考虑到生物保留性和批次校正性能,scGPT在总体指标中也排名靠前。
- 图2a:用scVI、Seurat和Harmony在不同数据集上对scGPT模型(Few shot)进行批集成后的细胞类型聚类性能测试。
- b:Few shot和从头训练的scGPT模型基因嵌入图谱的比较。每种细胞类型的高变基因被突出显示。在从头开始训练的scGPT模型中学习到的细胞嵌入的UMAP图被可视化见右。
- c:批次整合的定量评价。
细胞类型注释
细胞类型注释是在聚类之后的单细胞分析中至关重要的一步,它解决了测序组织中的异质性问题,并为进一步研究细胞和基因功能提供了基础,以获得生物学和病理学的见解。虽然已经有了几种细胞注释方法,如cellAssign、singleR和Chetah,但它们通常需要在模型输入之前进行降维,这可能会导致信息丢失。相比之下,scGPT的Transformer模型可以直接以无偏的方式接受基因表达数据作为输入,并对整个高变基因集进行全面的分析。这种方法提供了更高的可靠性和改进的细胞类型分类准确性。针对细胞类型注释任务,作者使用交叉熵损失对预训练的scGPT模型进行了微调,以便根据新的参考数据集的真实标签进行训练。以人胰腺细胞数据集为例,作者在参考集上训练了scGPT模型,并在不同的查询集上验证了分类性能。图3A和B显示了通过真实标签与预测的细胞类型着色的细胞特征,scGPT模型展现出高达96.7%的准确性得分。该模型在预测大多数细胞类型方面也表现出较高的精度,除了参考集中细胞数量极低的稀有细胞类型(见图3C)。

- 图3:细胞类型注释
多组学整合
单细胞多组学(scMultiomic)数据一次性提供了多个遗传调控视图,包括表观遗传学、转录组学和翻译活性。它为增强特征和细胞表示学习提供了丰富的机会。然而,挑战在于如何可靠地聚合来自多个视图的细胞表示,同时保留生物信号。
scGPT框架可以轻松扩展到集成多个测序数据模态。scMulti-omic数据中的每种组学类型(例如基因表达、染色质可及性和蛋白质丰度)类似于NLG中的不同语言。类似地,scGPT支持从不同测序模态联合优化多组学token。该框架还允许通过扩展"词汇表"无缝添加新的测序模态到现有的预训练网络中。在基准实验中,与现有的最先进方法相比,scGPT在细胞表示学习和多组学批次集成任务中展现出出色的性能(见图4)。
 文章来源:https://www.toymoban.com/news/detail-542132.html
文章来源:https://www.toymoban.com/news/detail-542132.html
- 图4:多组学整合
基因网络推断
转录因子、辅因子和靶基因之间的相互作用构成了基因调控网络(Gene Regulatory Network, GRN),介导着重要的生物过程。现有的GRN推断方法通常依赖于静态基因表达的相关性或伪时间估计作为因果图的替代。通过基因标记的生成式训练,scGPT隐式地编码了这些关系在其基因特征中。因此,可以应用scGPT展示从其基因特征网络中分组功能相关基因并区分功能不同基因的能力。在图5A中,作者展示了经过预训练的基因特征网络中人类白细胞抗原(Human Leukocyte Antigens, HLA)的相似性网络。在零样本设置下,scGPT模型突出显示了两个与两个经过充分研究的触发不同免疫反应的HLA类别对应的聚类,即HLA类I和HLA类II。HLA类I抗原HLA-A、C和E被CD8+ T细胞识别以介导细胞杀伤,而HLA类II抗原HLA-DR、DP和DQ被CD4+ T细胞识别以触发更广泛的辅助功能。对于在免疫人类数据集上进行微调的scGPT模型,作者探索了与该数据集中存在的免疫细胞类型特定的CD抗原网络(见图5C)。预训练的scGPT能够将CD3E、D和G基因识别为编码T细胞激活的T3复合物,将CD79A和B识别为B细胞信号传导,将CD8A和B识别为HLA类I分子的共受体。通过微调的scGPT进一步突出了CD36和CD14之间的关联,它们是单核细胞和巨噬细胞的marker。这证明了scGPT从预训练中学到的知识的泛化能力,并提取了与微调数据集相关的具体信息。 文章来源地址https://www.toymoban.com/news/detail-542132.html
文章来源地址https://www.toymoban.com/news/detail-542132.html
- 图5:基因网络推断
到了这里,关于基于GPT构建单细胞多组学基础模型的文章就介绍完了。如果您还想了解更多内容,请在右上角搜索TOY模板网以前的文章或继续浏览下面的相关文章,希望大家以后多多支持TOY模板网!