一、背景

我们选择下载直接可以使用的版本,右键保存链接地址,打开Linux系统(以Ubuntu为例)
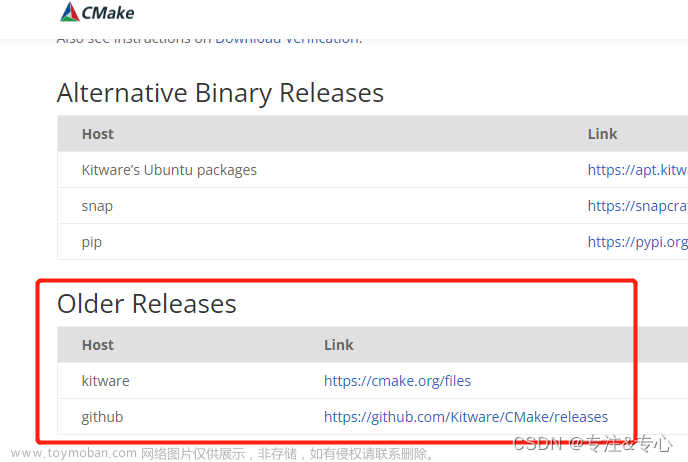
历史版本下载地址:Index of /files
注意:
这里不要装太老的cmake版本,因为后面make的时候没法做,要装 3.16 以上的

二、下载
创建/opt/cmake目录,在该目录下执行
wget -c https://cmake.org/files/v3.22/cmake-3.22.1-linux-x86_64.tar.gz
三、解压
tar xvf cmake-3.22.1-linux-x86_64.tar.gz
四、设置环境变量
vim /etc/profile
在文件最后增加两句
export CMAKE_HOME=/opt/cmake/cmake-3.5.2-Linux-x86_64/bin【注意这里要写你自己的cmake的bin地址】
export PATH=$CMAKE_HOME:$PATH
保存退出,刷新环境变量
source /etc/profile
检验是否安装成功
cmake -version
 文章来源:https://www.toymoban.com/news/detail-721728.html
文章来源:https://www.toymoban.com/news/detail-721728.html
Linux下安装CMake详细教程_上善若海的博客-CSDN博客_cmake安装教程linux文章来源地址https://www.toymoban.com/news/detail-721728.html
到了这里,关于Linux下安装 CMake的文章就介绍完了。如果您还想了解更多内容,请在右上角搜索TOY模板网以前的文章或继续浏览下面的相关文章,希望大家以后多多支持TOY模板网!